Welcome!
This community is for professionals and enthusiasts of our products and services. Share and discuss the best content and new marketing ideas, build your professional profile and become a better marketer together.
基于机器学习翻译,仅供参考。
练习2:查找CRISPR位点并检查脱靶匹配度
除了感兴趣的基因之外,gRNA序列可以结合基因组中的其他区域。Cas9也会影响这些基因组区域的序列。根据这些其他结合位点的位置,这可能会产生严重的意外后果。当选择CRISPR位点时,我们通常感兴趣的是基因组中gRNA序列可能与多少个其他位置结合以及这可能具有什么影响。Geneious中的Find CRISPR位点工具可以搜索非目标绑定,并使用此信息为每个CRISPR位点计算得分。
在第一个练习中,您使用Find CRISPR位点功能查找LYP1 CDS中的所有“GN(20)GG”位点。在本练习中,您还将检查异常与酿酒酵母基因组的结合情况。
要做到这一点,首先需要创建一个您希望针对脱靶结合位点进行测试的序列数据库 - 这通常是您感兴趣生物组织的整个基因组,但可以包含其他序列,例如靶向向量。要制作数据库,请在Geneious中创建一个新的空文件夹,然后将要测试的序列导入到该文件夹中。由于研究人员可能希望测试的基因组种类繁多,基因组序列可能非常大,并且可能会发布新版本的基因组组合,Geneious不包含任何全基因组序列的内置拷贝。可以使用NCBI直接从NCBI下载基因组位于Geneious的Sources面板底部的文件夹(这是Geneious左侧的面板)。常用研究的基因组(例如,人类,斑马鱼和大鼠基因组)可以从NCBI中下载,使用Geniouss文件夹中的Geneomes文件夹中的链接。基因组也可以从其他来源下载并使用通用文件格式导入Geneious。
在本教程中,已经为您创建了脱靶序列的数据库。由于本教程确定了酿酒酵母 LYP1基因中的CRISPR位点,我们对酿酒酵母基因组中的脱靶结合感兴趣 。酿酒酵母的完整基因组已从NCBI下载并置于酵母基因组文件夹中。要查看这些序列,请打开此文件夹。
要运行Find CRISPR位点工具来识别CRISPR位点并检查脱靶绑定,请选择LYP1 CDS文档(geneious的demo数据,网上提供下载)并转到克隆→查找CRISPR位点。
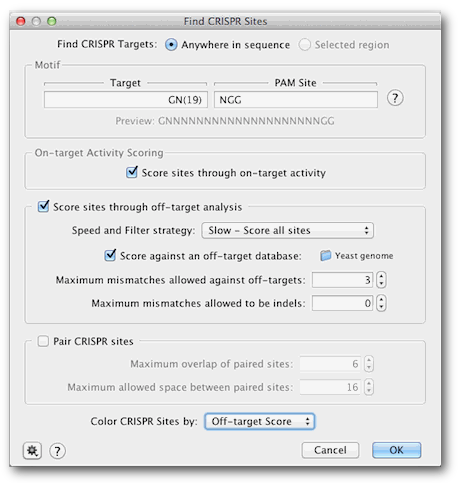
在Binding位点旁边,依次选择Anywhere。在目标类型“GN(19)” 旁边的字段以及PAM Site字段中键入“NGG”。通过检查目标活动保留分数位点。
现在通过脱靶分析和分数数据库来评分分数位点。然后选择包含我们想测试的序列的文件夹以进行非目标结合:点击选择一个文件夹并选择“酵母基因组”文件夹。对于速度和过滤策略,请选择慢速 - 对所有位点进行评分。将最大不匹配设置保留为缺省值3,以便与脱离目标不匹配,并允许0为indels。
注意:如果我们没有选择Score作为脱离目标数据库,但选择在较大序列的选定子区域内查找位点,Geneious将自动测试序列的未选定区域以进行脱靶结合。
这一次,我们希望以偏离目标分数为结果轨迹着色,因此请确保此选项在Color CRISPR sites by设置。对话框现在应该如下图所示。点击确定运行搜索。

CRISPR位点查找工具运行后,您将看到以下消息:
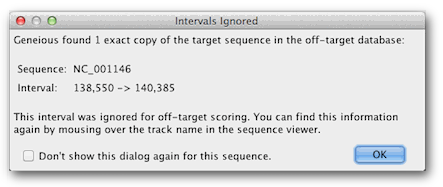

此消息出现是因为我们在LYP1 CDS中搜索CRISPR位点的序列也存在于脱靶数据库中,因为它是酵母基因组的一部分。由于它们很可能成为本身,因此该区域内的匹配被忽略,因为它们可能是向导位点guide site本身。
与之前的练习一样,Geneious在LYP1序列中注释了41个“GN(20)GG”CRISPR位点,并将它们归入CRISPR Sites。
序列中的每个CRISPR位点已根据其可能结合的脱靶位点数量以及脱靶位点与原序列的相似程度进行评分。“非目标分数”是根据麻省理工学院张峰实验室开发的方法计算出来的(欲了解更多信息,请点击这里)。根据每个脱离目标位点与原始CRISPR位点的相似程度以及发生何种不匹配(PAM位点附近的不匹配将比距离PAM位点更远的不匹配更多地影响绑定)给出评分。脱靶位点的较高分数表明与原始CRISPR位点更高的相似性(并且因此CRISPR / Cas复合物与脱靶标结合的可能性更高)。CRISPR位点的总分是100%减去目标基因组中偏离分数的加权总和。因此,更高的分数表示更好的CRISPR位点,而潜在的异地目标很少或很弱。
序列中的CRISPR注释现在根据其CRISPR分数进行着色。此配色方案使用红色到绿色的渐变色,CRISPR分数为20%时注释为纯红色,85%为纯黄色,100%时为纯绿色。
单击保存按钮将注释轨道保存在序列中。
打开注解选项卡。要仅显示注释表中的CRISPR注释,请单击“ 类型”并选择“ CRISPR”。单击列并勾选脱离目标分数和#脱离目标位点(这些可能已被选中)。这些列现在应该在注释表中可见。
单击注释表中列的名称将按该列中的值对表中的行进行排序。列名旁边将出现一个小三角形,指示行是从最小值到最大值进行排序,反之亦然。再次单击列名将颠倒行排序的方向。按照最低到最高的脱靶评分对注释表的行进行排序。
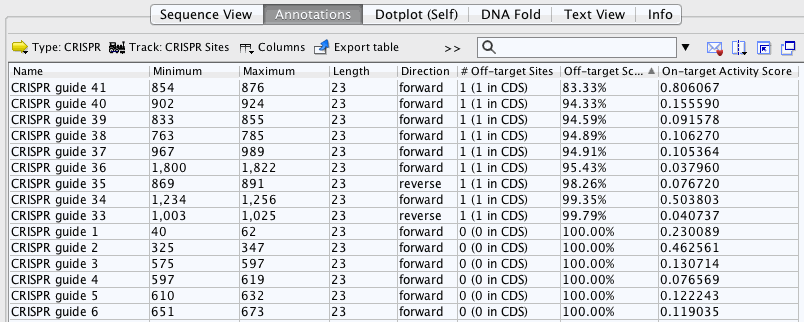

从这张表中你可以看到许多CRISPR位点的偏离目标得分为100%,这意味着它们没有脱离匹配的匹配符合无插入的条件和3个或更少的与gRNA错配(但是它们可能有额外的3个以上不匹配的脱靶点)。CRISPR guide 41具有最低的脱靶评分。在表格中选择此行并返回序列视图。现在应该在序列视图中选择“CRISPR guide 41”注释。将鼠标悬停在注释上,出现一个黄色的工具提示。此工具提示包含有关此CRISPR位点的更多信息。


该位点的CRISPR得分为83.33%。酿酒酵母基因组中只有一个此脱序结合位点,但它与CRISPR guide完全匹配,所以脱靶评分为100%。该位点位于136,903位的ALP1 CDS→14号染色体的136,925位。
对于具有多个脱离目标位点的CRISPR guide,只有前五位将列在工具提示中。此信息也可以从“ 注释”选项卡查看,排序和导出。
要从序列中删除这些注释,请单击序列视图中条目名称左侧的箭头,然后选择删除条目并单击保存按钮。
未完,继续[4]
随时了解最新的技术快讯
关于这个技术问答社区
问题工具
统计数据
| 问: 18-5-20 下午2:00 |
| 看到: 10065 时间 |
| 最后更新: 18-5-20 下午2:28 |


